URL: https://www.desy.de/aktuelles/news_suche/index_ger.html
Breadcrumb Navigation
[LgDashBoard2@/e409/e116959/e119238][getObjProperty]: key=title[py] Traceback (most recent call last): File "/home/zeoclients/parts/products/zms/_objattrs.py", line 679, in getObjProperty value = metaObjAttr['py'](zmscontext=self) File "/home/zeoclients/parts/zope2/lib/python/Shared/DC/Scripts/Bindings.py", line 313, in __call__ return self._bindAndExec(args, kw, None) File "/home/zeoclients/parts/zope2/lib/python/Shared/DC/Scripts/Bindings.py", line 350, in _bindAndExec return self._exec(bound_data, args, kw) File "/home/zeoclients/parts/zope2/lib/python/Products/PythonScripts/PythonScript.py", line 328, in _exec result = f(*args, **kw) File "Script (Python)", line 6, in LgDashBoard2.title UnicodeDecodeError: 'ascii' codec can't decode byte 0xc3 in position 1: ordinal not in range(128)
News-Suche
Meldungen vom Forschungszentrum DESY
Röntgenlaser entschlüsselt Stoppschalter für Zellsignale
Ein bislang unerreichter Einblick in die Steuerung biologischer Zellen ist Forschern mit dem weltstärksten Röntgenlaser in Kalifornien gelungen. Wie mit einer Art Supermikroskop haben die Wissenschaftler an der Linac Coherent Light Source (LCLS) des US-Forschungszentrums SLAC die genaue Funktionsweise eines zentralen Aus-Schalters für Zellsignale entschlüsselt. Die Erkenntnisse können den Weg zur Entwicklung zielgenauer Medikamente bahnen. Das internationale Forscherteam unter Beteiligung von DESY-Wissenschaftlern stellt seine Arbeit im britischen Fachjournal „Nature“ vor.
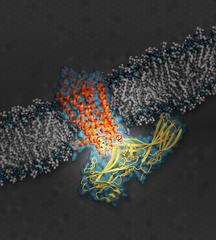
Die Illustration zeigt das Signalprotein Arrestin (gelb), angedockt Rhodopsin (orange), einen G-Protein-gekoppelten Rezeptor (GPCR). GPCRs spielen eine wichtige Rolle bei der Signalübertragung in Zellen. (Illustration: SLAC National Accelerator Laboratory)
Die Forscher um Prof. Eric Xu vom Van Andel Research Institute in den USA hatten das Sehpigment Rhodopsin untersucht, das in den Stäbchen der menschlichen Netzhaut vorkommt und für das Hell-Dunkel-Sehen zuständig ist. Es gehört zur großen Klasse der G-Protein-gekoppelten Rezeptoren (GPCR), über die rund jedes dritte Medikament seine Wirkung entfaltet – vom Schmerzmittel bis zum Blutdrucksenker.
Diese Rezeptoren übertragen Signale mit Hilfe der sogenannten Guanosintriphosphat-bindenden Proteine (G-Proteine). In der Übertragung von Signalen wie beispielsweise beim Sehen oder anderen nervlichen Aktivitäten ist das Abschalten eines Signals jedoch ebenso wichtig wie das Anschalten. Proteine einer anderen Familie, die Arrestine, können das Andocken der G-Proteine blockieren und die Signalübertragung damit abschalten. Auf diese Weise dienen Arrestine als wichtige Aus-Schalter in der Zellsteuerung.
Wie die Arrestine jedoch genau an die Rezeptoren ankoppeln, hat bislang noch niemand gesehen. Aus den Daten der Röntgenlaserexperimente haben die Wissenschaftler nun die erste dreidimensionale Aufnahme eines Arrestins gelungen, das an einen Rezeptor gebunden ist. „Diese Studie ist ein wichtiger erster Schritt“, urteilt Biochemie-Professor Jeffrey Benovic von der Thomas-Jefferson-Universität in Philadelphia, der nicht an der Untersuchung beteiligt war. Die Entschlüsselung der genauen Struktur an der Wechselwirkungsstelle der beiden Biomoleküle könne enorme Auswirkungen auf die Arzneientwicklung haben.
Denn viele der existierenden Medikamente, die über GPCR wirken, blockieren sowohl das Andocken von G-Proteinen als auch von Arrestinen. „Das neue Paradigma in der Medikamentenentwicklung lautet, einen zielgenauen Pfad zu finden, um entweder den Arrestin-Signalweg oder den G-Protein-Signalweg zu aktivieren und so eine bessere Wirkung zu erzielen“, erläutert Forschungsleiter Xu. Die Entschlüsselung der Arrestin-Kopplung an Rhodopsin ist dabei eine Vorlage, nach der sich nun auch die genaue Arrestin-Wirkung bei anderen GPCR untersuchen lassen könnte, wie Xu hofft.
Für die Studie hatten die Forscher zunächst winzige Kristalle aus verbundenen Rhodopsin-Arrestin-Komplexen gezüchtet. Die atomare Struktur solcher Kristalle lässt sich darüber ermitteln, wie sie Röntgenstrahlung streuen. Diese Röntgenkristallographie ist heute ein Standardverfahren in der Strukturbiologie und in zahlreichen anderen Disziplinen.
„Biomoleküle lassen sich allerdings meist nur sehr widerstrebend in Kristallform zwingen, weil dies ihrer natürlichen Funktion widerspricht“, erläutert DESY-Forscher Dr. Anton Barty vom Center for Free-Electron Laser Science, der an der Datenauswertung intensiv mitgearbeitet hat. Für die Rhodopsin-Arrestin-Untersuchung mussten die Wissenschaftler sich mit Mikrokristallen zufriedengeben, die lediglich tausendstel Millimeter klein waren. „Solche Mikrokristalle sind normalerweise sehr schwer zu untersuchen, aber mit dem hellen Röntgenlicht der LCLS gelangen von fast 19 000 dieser Mikrokristalle so gute Streubild-Aufnahmen, dass sich aus der Kombination dieser Streubilder die Struktur des Molekülkomplexes berechnen ließ“, berichtet Barty.
Diese Methode der seriellen Femtosekunden-Röntgenkristallographie (SFX) wurde maßgeblich von DESY-Forscher Prof. Henry Chapman und seinem Team entwickelt. „Die neue Arbeit zeigt das enorme Potenzial von Röntgenlasern für die Strukturaufklärung“, sagt Chapman. Der hier untersuchte Molekülkomplex war der bisher schwierigste, der so entschlüsselt wurde, und stellt damit die Schlagkraft der Methode unter Beweis. Mit dem European XFEL bauen DESY und zehn internationale Partner gegenwärtig einen Röntgenlaser der nächsten Generation, der voraussichtlich 2017 den Forschungsbetrieb beginnen soll.
An der Untersuchung waren außer dem Van Andel Research Institute, SLAC und DESY auch die Arizona State University, die University of Southern California, die National University of Singapore, das New York Structural Biology Center, The Scripps Research Institute, die University of California in Los Angeles, die University of Toronto in Kanada, die Vanderbilt University, das Pekinger Computational Science Research Center in China, die University of Wisconsin-Milwaukee, die Chinesische Akademie der Wissenschaften, das Paul-Scherrer-Institut in der Schweiz, das Trinity College in Irland, die University of Chicago und die Universität Konstanz beteiligt.
Originalarbeit:
„Crystal structure of rhodopsin bound to arrestin by femtosecond X-ray laser“; Yanyong Kang, X. Edward Zhou, Xiang Gao, Yuanzheng He et al.; „Nature“ (2015); DOI: 10.1038/nature14656



